中国农业科学院博士后利用泛串联重复序列变异实现水稻农艺性状精准调控
近日,中国农业科学院深圳农业基因组研究所(岭南现代农业科学与技术广东省实验室深圳分中心)联合崖州湾国家实验室、河南大学、福建省农科院等单位在《Nature Communications》上在线发表了题为“The pan-tandem repeat map highlights multiallelic variants underlying gene expression and agronomic traits in rice”的研究论文。研究基于图形超级泛基因组,构建了群体水平高精度的水稻串联重复(Tandem Repeat,TR)变异数据集,全面评估了TR变异的遗传特征,系统解析了TR变异对基因表达和表型的影响,并以实例证明了TR变异精准调控水稻复杂农艺性状的应用潜力。该研究丰富了水稻育种的可用变异库,为水稻种质资源的高效挖掘和农艺性状精准设计提供了重要资源。
水稻是世界上最重要的粮食作物之一,水稻品种的不断改良得益于对种质资源的利用和挖掘,尤其是优异基因挖掘与利用。全球水稻种质资源丰富的遗传多样性为水稻有利性状的遗传改良提供了重要基础。因此,不断推动解析水稻群体基因组多样性,深入挖掘水稻优异自然变异对于水稻育种改良具有重要意义。
围绕水稻优异基因挖掘与利用,该课题组前期开展了系列工作。为了弥补参考基因组信息不完整造成的自然变异丢失,提升优异基因挖掘的准确度,团队联合多家单位完成了水稻首个日本晴完整参考基因组组装和高质量注释(Shang et al., Molecular Plant, 2023),为了充分挖掘种质资源群体优异等位基因,团队联合多家单位利用全球水稻核心种质资源构建了包含野生稻和栽培稻的水稻超级泛基因组,整合了稻属水平的群体基因组自然变异多样性,使更多的包含转座子变异、倒位变异、着丝粒区等复杂区域隐藏的优异自然变异能够被高效鉴定和挖掘(Shang et al., Cell Research, 2022; Lin et al., Journal of Integrative Plant Biology, 2023;Li et al., National Science Review, 2024;He et al., Science Bulletin, 2024)。为了挖掘种质资源群体中稀有优异等位基因,团队利用超级泛基因组对万份规模水稻群体基因组变异进行了全面解析,大幅提升了对稀有优异自然变异的挖掘和应用能力(Wang et al., Nucleic Acids Research, 2023)。为了进一步拓宽和挖掘水稻种质资源中可用的遗传资源,对包含TR在内的复等位变异的群体遗传学应用开始逐步引起重视。
TRs是DNA中以两个或多个核苷酸为一组重复且相邻出现的一段序列。TR往往具有高度多态性,存在多种复等位形式。但前期研究中TRs一般被当做遗传标记进行QTL定位,TR本身对基因表达和表型的研究较少。该研究利用231份材料构成的水稻图形超级泛基因组进行TR变异位点注释,发现了227,391个复等位的TR变异位点,其中54,416个位点在参考基因组日本晴上以非串联重复形式出现。仅1/3的TR变异与周围的二等位变异(如SNPs、Indels、PAVs)存在强连锁。用202份水稻叶片群体转录组和193份穗群体转录组数据,该研究系统分析了TRs变异和水稻基因表达的关联。通过精细定位和控制变量分析,发现排除二等位变异影响后,485和511个TR变异仍然独立影响穗和叶片基因表达。以其中两个基因为例,该研究通过基因编辑TR位点,验证了TR变异对基因表达和表型的影响,展示了TRs变异在精准育种中的应用潜力。
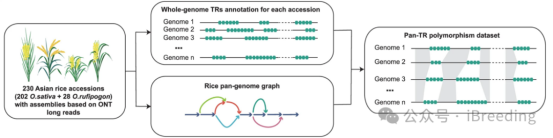
图1 图形化泛串联重复变异图谱构建过程示意图。
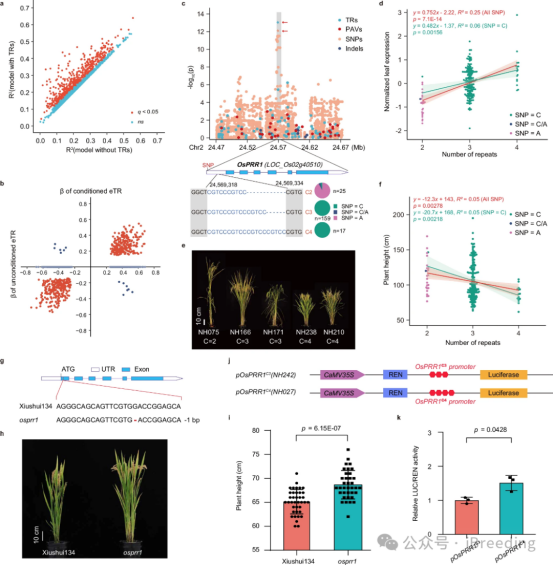
图2 通过精细定位解析 OsPRR1 启动子TRs变异对基因表达和表型的影响。
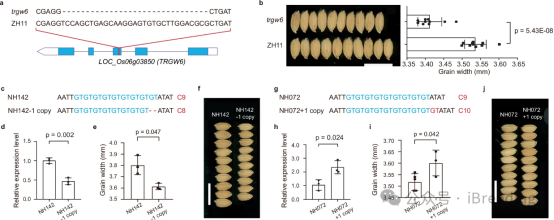
图3 通过基因编辑TR区域实现水稻精准育种。
中国农业科学院深圳农业基因组研究所(岭南现代农业科学与技术广东省实验室深圳分中心)商连光研究员和崖州湾国家实验室钱前院士为论文的共同通讯作者。基因组所副研究员贺慧英、博士后冷月、基因组所与河南大学联培硕士生曹兴岚和福建省农科院朱义旺博士为论文共同第一作者。该研究得到国家自然科学基金基础科学中心、广东省自然科学基金杰出青年基金、中国农业科学院科技创新工程科学中心和中国农科院青年创新专项资金资助。该工作得到了基因组所、中国水稻所和崖州湾科技城超级计算平台的支持。

